NiPorph Enthalpie der Komplexe mit verschiedenen Liganden: Unterschied zwischen den Versionen
KKeine Bearbeitungszusammenfassung |
|||
| Zeile 18: | Zeile 18: | ||
=== Vergleich mit experimentellen Daten und Deutung (fürs paper) === | === Vergleich mit experimentellen Daten und Deutung (fürs paper/supp. material) === | ||
verwendete Rechenmethode: ri-PBE/DZP (+ marij) - Turbomole6.0 | verwendete Rechenmethode: ri-PBE/DZP/ri-universal (+ marij) - Turbomole6.0 | ||
Es ist bekannt, dass gerade für das vorliegende Problem Dispersionswechselwirkungen eine wesentliche Rolle für die Stärke und Länge der N-Ni Bindungen Komplexbindungen zwischen Pyridin und Ni-Porphyrin spielen, die durch reine Dichtefunktionalmethoden nicht wiedergegeben werden. | Es ist bekannt, dass gerade für das vorliegende Problem Dispersionswechselwirkungen eine wesentliche Rolle für die Stärke und Länge der N-Ni Bindungen Komplexbindungen zwischen Pyridin und Ni-Porphyrin spielen, die durch reine Dichtefunktionalmethoden nicht wiedergegeben werden.[Honza Abstract] | ||
Eine empirische Korrektur der Dispersionswechselwirkung ist im Programm | Eine empirische Korrektur der Dispersionswechselwirkung[Grimme] ist im Programm Turbomole6.0 implementiert, der Einsatz bei der vorliegenden Substanzklasse führte in Vorstudien aber dazu, dass sich die aromatischen Reste "aufeinanderlegten" und damit die Gesamtstruktur der mit Dispersionsww. optimierten Komplexe nicht mehr den erhaltenen Röntgenstukurdaten[Steffens cyanopy x-ray],'''[entweder eine der alten Strukturen oder cyanopy+disp aus alten Kippstrukturen optimieren]''' mit aufrecht stehenden Pyridinliganden entsprachen. | ||
Während dies in vacuo wahrscheinlich tatsächlich der Komplexstruktur entsprechen würde, liegen in Toluol als Lösemittel genügend Möglichkeiten für Kompensation der Dispersionsww. zwischen aromatischen Resten durch das Lösemittel vor. Aus diesem Grunde wurden die Optimierungen ohne Dispersionskorrektur durchgeführt. | Während dies in vacuo wahrscheinlich tatsächlich der Komplexstruktur entsprechen würde, liegen in Toluol als Lösemittel genügend Möglichkeiten für Kompensation der Dispersionsww. zwischen aromatischen Resten durch das Lösemittel vor. Aus diesem Grunde wurden die Optimierungen ohne Dispersionskorrektur durchgeführt, auch wenn dies bedeutet, dass die Pyriding-N · Ni Bindung systematisch fehlerbehaftet modelliert wird. | ||
Beim Vergleich mit den | Beim Vergleich der theoretisch ehrhaltenen Werten mit den experimentellen Daten fällt auf, dass die Komplexationsenthalpien der 1:1 Komplexe grundsätzlich recht gut übereinstimmen (ca. 1 kcal/mol Abweichung max.). Systematisch signifikant größer (5-6 kcal/mol) sind die erhaltenen Komplexationsenthalphien der 2:1 Komplexe in der Rechnung im Vergleich zum Experiment. Die Erklärung dafür liegt unserer Meinung nach in der Vernachlässigung der Dispersionsww in der Rechnung. | ||
- Vergleich d(N·Ni) x-ray/'''disp'''/nodisp cyanopy 2:1 '''(TODO)''' | |||
- Struktur (1:1 mit Disp./ohne) | |||
Wie man sieht ragt beim 1:1 Komplex | |||
Es scheint, dass die Komplexationsenthalpien grundsätzlich auf dem Niveau | |||
[[Kategorie:Akherges]] | [[Kategorie:Akherges]] | ||
Version vom 23. September 2009, 09:30 Uhr
Ni-Porphyrin Komplexe mit Stickstoffbasen
Nickelporphyrin
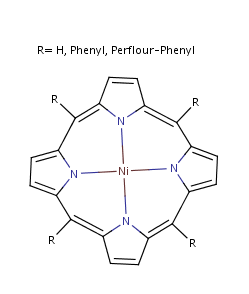
sämtliche Rechnungen hier mit R=Perflourphenyl
Liganden
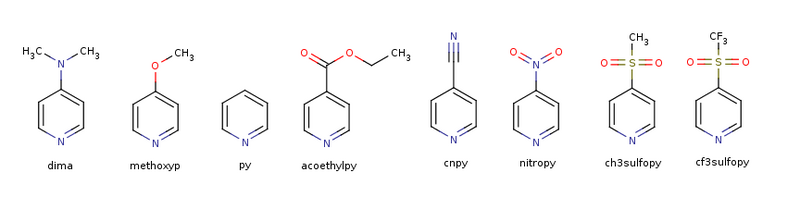
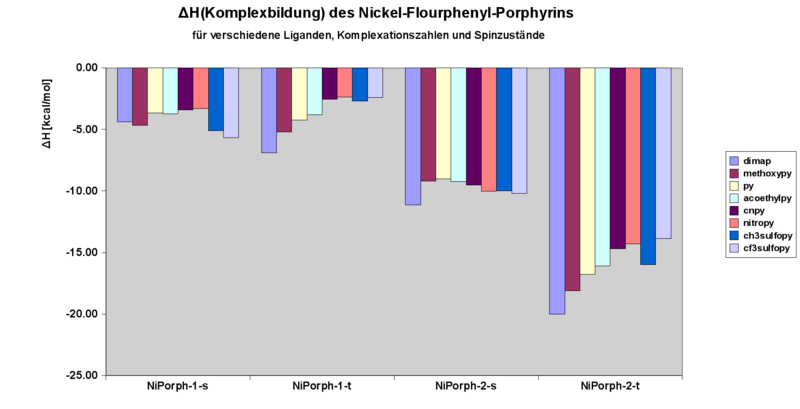
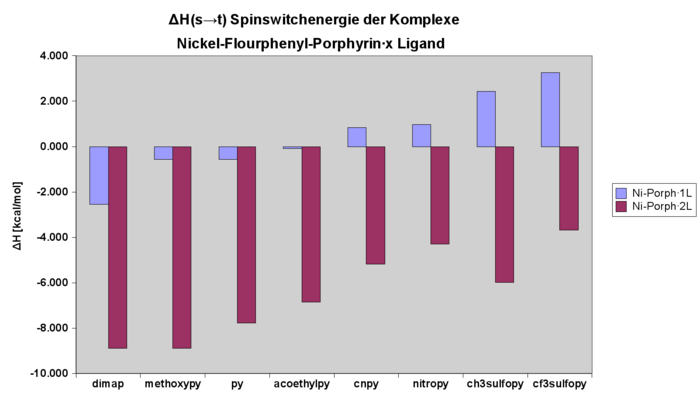
Nickel-Porphyrin Komplexe (NiPorph ∙ N Ligand, N=0,1,2):
sämtliche Werte gerechnet bei 298.15K
Enthalpie der Komplexation
Enthalpie des Spinswitches
Vergleich mit experimentellen Daten und Deutung (fürs paper/supp. material)
verwendete Rechenmethode: ri-PBE/DZP/ri-universal (+ marij) - Turbomole6.0 Es ist bekannt, dass gerade für das vorliegende Problem Dispersionswechselwirkungen eine wesentliche Rolle für die Stärke und Länge der N-Ni Bindungen Komplexbindungen zwischen Pyridin und Ni-Porphyrin spielen, die durch reine Dichtefunktionalmethoden nicht wiedergegeben werden.[Honza Abstract]
Eine empirische Korrektur der Dispersionswechselwirkung[Grimme] ist im Programm Turbomole6.0 implementiert, der Einsatz bei der vorliegenden Substanzklasse führte in Vorstudien aber dazu, dass sich die aromatischen Reste "aufeinanderlegten" und damit die Gesamtstruktur der mit Dispersionsww. optimierten Komplexe nicht mehr den erhaltenen Röntgenstukurdaten[Steffens cyanopy x-ray],[entweder eine der alten Strukturen oder cyanopy+disp aus alten Kippstrukturen optimieren] mit aufrecht stehenden Pyridinliganden entsprachen.
Während dies in vacuo wahrscheinlich tatsächlich der Komplexstruktur entsprechen würde, liegen in Toluol als Lösemittel genügend Möglichkeiten für Kompensation der Dispersionsww. zwischen aromatischen Resten durch das Lösemittel vor. Aus diesem Grunde wurden die Optimierungen ohne Dispersionskorrektur durchgeführt, auch wenn dies bedeutet, dass die Pyriding-N · Ni Bindung systematisch fehlerbehaftet modelliert wird.
Beim Vergleich der theoretisch ehrhaltenen Werten mit den experimentellen Daten fällt auf, dass die Komplexationsenthalpien der 1:1 Komplexe grundsätzlich recht gut übereinstimmen (ca. 1 kcal/mol Abweichung max.). Systematisch signifikant größer (5-6 kcal/mol) sind die erhaltenen Komplexationsenthalphien der 2:1 Komplexe in der Rechnung im Vergleich zum Experiment. Die Erklärung dafür liegt unserer Meinung nach in der Vernachlässigung der Dispersionsww in der Rechnung.
- Vergleich d(N·Ni) x-ray/disp/nodisp cyanopy 2:1 (TODO) - Struktur (1:1 mit Disp./ohne)
Wie man sieht ragt beim 1:1 Komplex Es scheint, dass die Komplexationsenthalpien grundsätzlich auf dem Niveau